我们的基因体时代-AI, Data和生物资讯 Day24- 使用tidyverse观念来分析基因资料:plyranges
上一篇我们的基因体时代-AI, Data和生物资讯 Day23- 基因注释资料在Bioconductor中视觉化之呈现:Gviz我们示范如何将前几天的知识整合一起,先使用AnnotationHub来取用人类基因组相关的资料包(org.Hs.eg.db, TxDb.Hsapiens.UCSC.hg38.knownGene, TxDb.Hsapiens.),再将这些资料转换成Bioconductor中用来分析基因资讯的资料结构IRanges, GRanges,最後使用Gviz视觉化来比较旧版本和新版本中RhD基因的资料差异。
接着我们再往下如何用tidyverse的资料科学语法来分析前几篇的基因资料,其实在前几天的代码中就使用蛮多tidyverse原生的library,但在处理IRanges和GRanges时却还是非tidyverse的写法,相对地不太顺畅和直觉,也许不是每个人会使用R语言的人都听过tidyverse,这边先稍微介绍一下。

R语言在这10年多其实越来越流行,虽然这三年在深度学习风潮後比较退火外,在资料科学家间其实是蛮好用的一个语言,一方面跟他友善资料处理有关,R语言这十年来如此受欢迎,第一个是其原生是由统计学家们所开发的程序语言,所以很多统计工具都有相关的程序包,第二个则是语法上的进步和友善的社群,很多不错的工具像是ggplot2, dplyr等等,算是让人很容易就上手,且可以很functional programming来做分析,而统计学家Hadley Wickham便是这几个重要library的开发者,其陆续形成了一个很棒的开发社群,同时还有商业化的公司Rstudio,提供不断进步的R语言编辑器,其中在Hadley Wickham的领导下,陆陆续续把资料分析会用到的工具大抵上都开发完善如dplyr, readr, stringr, purrr, tidyr, tibble等,最後他把这些整合到一个包,叫做tidyverse,这样一次就可以满足百分之八十以上的资料分析需求,在这个开发体系中有几个蛮好的原则,可参考这篇由Hadley Wickham撰写的文章The tidy tools manifesto:
- 重复使用已经使用的资料结构
- 将简单的函数整合到pipe中
- 拥抱函数编程(functional programming)的快感
- 为人类服务
导入tidyverse思维的基因资料处理包:plyranges
这个包的开发者Michael Lawrence其实就是当初开发IRange和GenomicRanges的人,所以可以理解他也会是把IRanges/GenomicRanges结构语法跟tidyverse整合的最佳人选,这个包建立的一些跟tidyverse一致的语法及跟基因资料处理相关的部分整合如下:
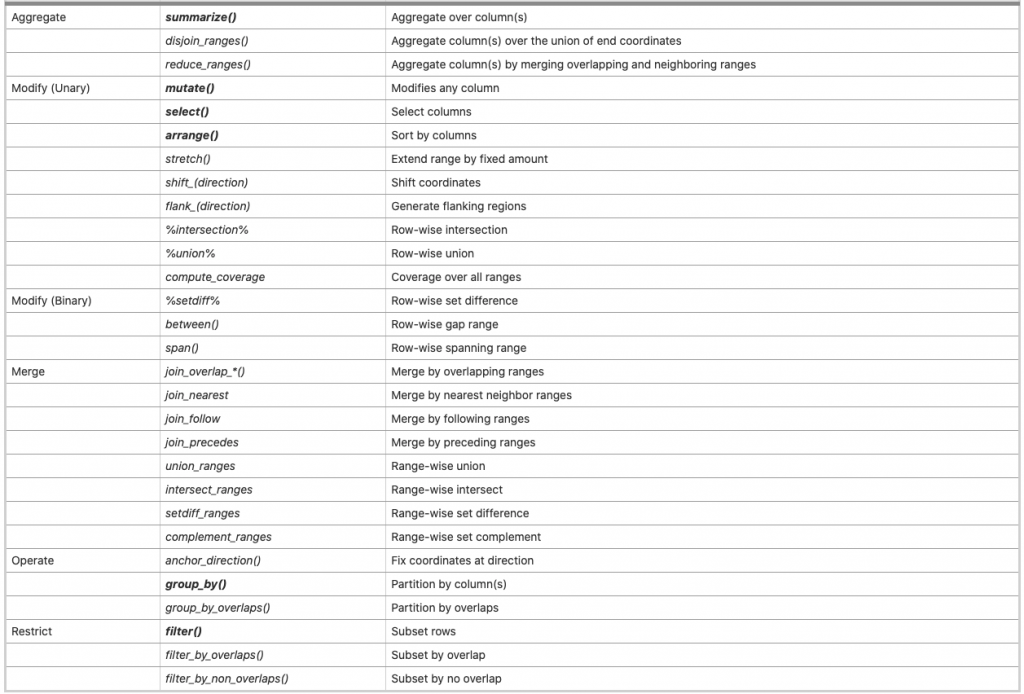
上面表格黑色粗体的部分就是沿袭自tidyverse中dplyr的语法:mutate, filter, summarize, groupy_by, select, arrange,其他则是跟IRange/GRange处理特异性相关的操作。
这边承袭昨天的代码,先取得多个跟输血医学相关的基因资讯:
library(org.Hs.eg.db)
library(TxDb.Hsapiens.UCSC.hg38.knownGene)
TxDb.hg38 <- TxDb.Hsapiens.UCSC.hg38.knownGene
gene.inform.df <- AnnotationDbi::select(org.Hs.eg.db,
keys = c("RHD", "VWF", "ADAMTS13"),
keytype = c("SYMBOL"),
columns = c("ENTREZID"))
hg38.cd.range.inform.df <- AnnotationDbi::select(TxDb.hg38,
keys = gene.inform.df$ENTREZID,
keytype = c("GENEID"),
columns = c("CDSCHROM", "CDSSTART","CDSID", "CDSEND", "CDSID","CDSSTRAND"))
使用as_iranges, as_granges转换物件
相对於使用IRanges或是GRanges来建立物件,plyranges提供一个接近tidyverse语法的作法,使用as_ranges和as_granges函数,就能将data.frame的资料,转换成IRanges和GRanges资料物件,但要遵守几个原则,要转换为iranges的data.frame,栏位至少要有start和end两栏,而使用as_grangrs转换为GRanges的物件,其data.frame至少要有三栏:start, end, seqnames,名字要一模一样,就可以成功转换,的确跟昨天和前天的语法比较一下,整个轻松简单许多。
# 使用as_iranges
blood.irgs <- hg38.cd.range.inform.df %>%
dplyr::select(CDSSTART, CDSEND) %>%
dplyr::rename("start"=CDSSTART, 'end'=CDSEND) %>%
as_iranges()
# 使用as_granges
blood.grgs <- hg38.cd.range.inform.df %>%
dplyr::select(CDSCHROM, CDSSTART, CDSEND, GENEID, CDSSTRAND) %>%
dplyr::rename("start"=CDSSTART, 'end'=CDSEND, 'seqnames'=CDSCHROM) %>%
as_granges()
>blood.irgs
IRanges object with 139 ranges and 0 metadata columns:
start end width
<integer> <integer> <integer>
[1] 25272548 25272695 148
[2] 25284573 25284759 187
[3] 25290641 25290791 151
[4] 25300946 25301093 148
[5] 25301520 25301686 167
... ... ... ...
[135] 255794 256004 211
[136] 281379 281529 151
[137] 256126 256195 70
[138] 259472 259511 40
[139] 282207 282309 103
>blood.grgs
GRanges object with 139 ranges and 2 metadata columns:
seqnames ranges strand | GENEID CDSSTRAND
<Rle> <IRanges> <Rle> | <character> <character>
[1] chr1 25272548-25272695 * | 6007 +
[2] chr1 25284573-25284759 * | 6007 +
[3] chr1 25290641-25290791 * | 6007 +
[4] chr1 25300946-25301093 * | 6007 +
[5] chr1 25301520-25301686 * | 6007 +
... ... ... ... . ... ...
[135] chr9_KN196479v1_fix 255794-256004 * | 11093 +
[136] chr9_KN196479v1_fix 281379-281529 * | 11093 +
[137] chr9_KN196479v1_fix 256126-256195 * | 11093 +
[138] chr9_KN196479v1_fix 259472-259511 * | 11093 +
[139] chr9_KN196479v1_fix 282207-282309 * | 11093 +
-------
seqinfo: 4 sequences from an unspecified genome; no seqlengths
直接使用filter来筛选IRanges/GRanges中的资讯
这边我觉得超级方便,可以直接导入dplyr中filter的语法,针对任一个column的数值做筛选,可以是数值上的大小,或是字串上面的异同,比如我可以使用在IRanges的资料上,筛选出只有片段大於200的序列,同样的,在比较资料复杂的GRanges物件,则可以做出如只要为在特定染色体上,或是後面meta data栏位的资讯,只是它的速度比想像中还慢,所以在较大的资料量下,可能要稍微等比较久一点。
> blood.irgs %>% filter(width > 200)
IRanges object with 17 ranges and 0 metadata columns:
start end width
<integer> <integer> <integer>
[1] 25328898 25329136 239
[2] 6110374 6110582 209
[3] 6075335 6075551 217
[4] 6056857 6057072 216
[5] 6052543 6052783 241
... ... ... ...
[13] 133429700 133429910 211
[14] 280509 280713 205
[15] 281379 281697 319
[16] 285068 285274 207
[17] 255794 256004 211
> blood.grgs %>% filter(GENEID == '6007')
GRanges object with 15 ranges and 2 metadata columns:
seqnames ranges strand | GENEID CDSSTRAND
<Rle> <IRanges> <Rle> | <character> <character>
[1] chr1 25272548-25272695 * | 6007 +
[2] chr1 25284573-25284759 * | 6007 +
[3] chr1 25290641-25290791 * | 6007 +
[4] chr1 25300946-25301093 * | 6007 +
[5] chr1 25301520-25301686 * | 6007 +
... ... ... ... . ... ...
[11] chr1 25317000-25317052 * | 6007 +
[12] chr1 25328898-25328924 * | 6007 +
[13] chr1 25321889-25321962 * | 6007 +
[14] chr1 25328898-25329136 * | 6007 +
[15] chr1 25328898-25329015 * | 6007 +
-------
seqinfo: 4 sequences from an unspecified genome; no seqlengths
修改IRanges/GRanges物件使用mutate
可以直接使用mutate来修改物件中的栏位,而其他栏位会同时调整,比如你修改这个基因区段的长度,那麽你则可已修改width,则start或end的位置会调整,可以使用anchor_start, anchor_end, anchor_centre, anchor_5p, anchor_3p来指定这样的调整,要以哪边为基准。
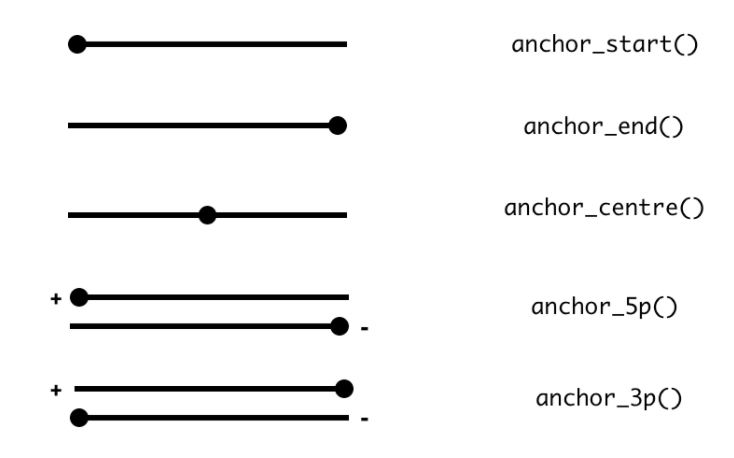
可以看一下面的示范:
> blood.irgs %>% mutate(width=10)
IRanges object with 139 ranges and 0 metadata columns:
start end width
<integer> <integer> <integer>
[1] 25272548 25272557 10
[2] 25284573 25284582 10
[3] 25290641 25290650 10
[4] 25300946 25300955 10
[5] 25301520 25301529 10
... ... ... ...
[135] 255794 255803 10
[136] 281379 281388 10
[137] 256126 256135 10
[138] 259472 259481 10
[139] 282207 282216 10
> blood.irgs %>% anchor_end() %>% mutate(width=10)
IRanges object with 139 ranges and 0 metadata columns:
start end width
<integer> <integer> <integer>
[1] 25272686 25272695 10
[2] 25284750 25284759 10
[3] 25290782 25290791 10
[4] 25301084 25301093 10
[5] 25301677 25301686 10
... ... ... ...
[135] 255995 256004 10
[136] 281520 281529 10
[137] 256186 256195 10
[138] 259502 259511 10
[139] 282300 282309 10
> blood.irgs %>% anchor_start() %>% mutate(width=10)
IRanges object with 139 ranges and 0 metadata columns:
start end width
<integer> <integer> <integer>
[1] 25272548 25272557 10
[2] 25284573 25284582 10
[3] 25290641 25290650 10
[4] 25300946 25300955 10
[5] 25301520 25301529 10
... ... ... ...
[135] 255794 255803 10
[136] 281379 281388 10
[137] 256126 256135 10
[138] 259472 259481 10
[139] 282207 282216 10
> blood.irgs %>% anchor_center() %>% mutate(width=10)
IRanges object with 139 ranges and 0 metadata columns:
start end width
<integer> <integer> <integer>
[1] 25272617 25272626 10
[2] 25284661 25284670 10
[3] 25290711 25290720 10
[4] 25301015 25301024 10
[5] 25301598 25301607 10
... ... ... ...
[135] 255894 255903 10
[136] 281449 281458 10
[137] 256156 256165 10
[138] 259487 259496 10
[139] 282253 282262 10
除了基础的建立物件和筛选,其实还能做超多方便的运算这部分真的蛮不错的!
Lee, S., Cook, D. & Lawrence, M. plyranges: a grammar of genomic data transformation. Genome Biol 20, 4 (2019). https://doi.org/10.1186/s13059-018-1597-8
Chapter 16. Visualizing Genomic Data Using Gviz and Bioconductor. Statistical Genomics. 2016
Ou J, Zhu LJ (2019). “trackViewer: a Bioconductor package for interactive and integrative visualization of multi-omics data.” Nature Methods, 16, 453–454. doi: 10.1038/s41592-019-0430-y, https://doi.org/10.1038/s41592-019-0430-y.
这个月的规划贴在这篇文章中我们的基因体时代-AI, Data和生物资讯 Overview,也会持续调整!我们的基因体时代是我经营的部落格,如有对於生物资讯、检验医学、资料视觉化、R语言有兴趣的话,可以来交流交流!
>>: [Day 19] 突如其来的需求变更!来聊函数式编程
Ruby on Rails Route 起步走
举个例⼦来说,这个网址: http://rubyonrails.com/posts/123 Rail...
卡夫卡的藏书阁【Book7】- Kafka 实作新增 Topic
「不要屈服,不要淡化,不要使它看来合逻辑,不要依据潮流而修改你的灵魂。相反的,狠狠的追随你最强烈的...
AutoML应用分享║DataRobot 年度线上大会 2021
一年一次关注全球AI实践家怎麽想 不知道大家习不习惯注册业界大厂办的活动? 撇除我们自己是DataR...
拆掉 v-model + computed get/set 到 vuex
为什麽要特别写 Form 表单攻略呢? 因为这是使用者可以输入资料的常见途径,一种可以「写入」资料的...
Day24 生产环境需要注意的部分
在介绍过监控、yaml 控管、网路的端点暴露与附载平衡後,官方有给我们一些在,生产环境部署的建议。透...