Python 演算法 Day 16 - Unsupervised
Chap.II Machine Learning 机器学习
https://yourfreetemplates.com/free-machine-learning-diagram/
Part 6. Unsupervised 非监督式学习
本篇会着重在先前提过的非监督式学习上。
它是机器学习的一种方法,在没有标记的训练集中,让学习器对输入的资料进行分类。
非监督学习主要藉由:聚类分析(cluster analysis)、关联规则(association rule)、维度缩减(dimensionality reduce)达成最终的分群。
而本篇会着重在 Cluster analysis 聚类分析上。
Cluster analysis 聚类分析
把相似的物件通过静态分类成不同的组别或者更多的 subset,让同子集中的物件都有相似属性。
依照分类方式,又分为:
Hard Clustering:数据集中的样本都被「100%」分到某一类别中。
Soft Clustering:数据集中的样本以「一定的概率」被分到某一类别中。
6-1. K-means Clustering K-平均聚类
即是一种硬聚类。透过不停迭代求解,直至分类到指定聚类数量,又称 EM 分析。
Expectation-step: 求得每个聚类的重心(mean)
Maximization-step: 把资料点归属给距离最近的聚类重心。
详细步骤为:
- 指定 k 值,把资料集随机拆成 k 个聚类。
- 以平均方式找出 k 个聚类重心(centroids),把资料点归属到距离最近的聚类中。
- 以新聚类平均找出新重心,重新对资料划分归属
- 重复 2~3 直到没有资料点改变聚类归属。
打个比方:假设有一资料集 (5,20,11,5,3,19,30,3,15)

至此,所有资料点不再更动,即完成。
但 k-means 的缺点如下:
- 以初始聚类重心确立初始划分并对其优化。初始聚类重心的选择对聚类结果有很大影响。
- k 值选定困难,实例中常难以估计数据集该分成多少聚类最合适。
- 因需循环迭代,面对大数据时会耗费庞大资源。
A. K-means++
K-means++ 改善了初始重心选取不当的问题。核心思想:初始的聚类重心间距要尽可能远。
步骤如下:
- 从输入的数据集中,随机选一个点作第一个聚类中心。
- 计算数据集的每一个点 x 与已选择的聚类中心的距离 D(x)
- D(x) 越大的点,被选作下一个新聚类中心的概率越大
- 重复 2~3,直到 k 个聚类中心被选出来
- 用这 k 个初始的聚类中心来执行 k-means 算法
以 make_blobs 创建的随机乱数为例:
from sklearn.datasets import make_blobs
X, y_true = make_blobs(
n_samples=300,
centers=4,
cluster_std=0.6,
random_state=0
)
from sklearn.cluster import KMeans
km = KMeans(
n_clusters=4,
random_state=0
)
km.fit(X)
y_pred = km.predict(X)
参数:
1. n_clusters: 即 k 值。默认 8。
2. max_iter: 执行一次 k-means 算法所进行的最大迭代数。默认 300。
3. n_init: k-means 算法会随机运行 n 次,将最好的聚类作初始化的结果。默认 10。
4. verbose: 列出优化过程。默认 0。
5. init: 初始化方法,有 ‘k-means++’(默认), ‘random’ 或 nd-array。
*更多的超参数可参考:https://www.twblogs.net/a/5b8aaade2b71775d1ce86b48*
y_true[:20], y_pred[:20]
>> (array([1, 3, 0, 3, 1, 1, 2, 0, 3, 3, 2, 3, 0, 3, 1, 0, 0, 1, 2, 2]),
array([0, 2, 1, 2, 0, 0, 3, 1, 2, 2, 3, 2, 1, 2, 0, 1, 1, 0, 3, 3]))
此时发现很多资料「看似」被预测错误,为什麽?
答:因为此为「非监督式学习」,没有 label,自然就不会依照我们所想的方式分类。
若我们把不同类别的 index 列举出来比较,会发现其实它们被分配到同个群:
import pandas as pd
true = pd.Series(y_true)
pred = pd.Series(y_pred)
print(true[true==1].index)
print(pred[pred==0].index)

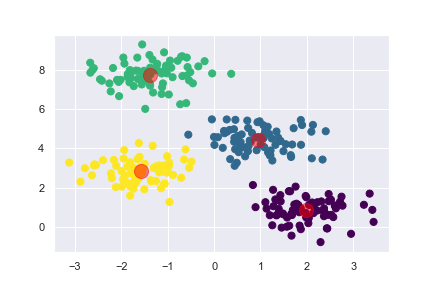
# 在图上标示重心
plt.scatter(X[:, 0], X[:, 1], c=y_pred, s=50, cmap='viridis')
centers = km.cluster_centers_
plt.scatter(centers[:, 0], centers[:, 1], c='red', s=200, alpha=0.5)
B. 决定聚类数目
解决了初始聚类重心的难题後,接着我们来解决 k 值选定。
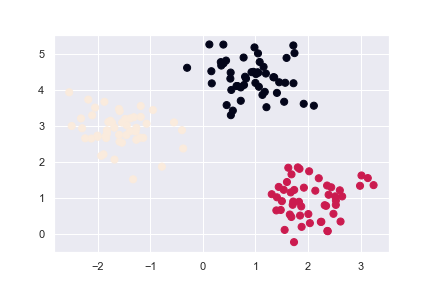
使用 make_blobs 产生的资料作以下两种范例:
# 图片美化用
import seaborn as sns; sns.set()
from sklearn.datasets import make_blobs
X, y_true = make_blobs(
n_samples=150,
n_features=2,
centers=3,
cluster_std=0.5,
shuffle=True,
random_state=0
)
plt.scatter(X[:, 0], X[:, 1], c=y_true, s=50)
B1. Elbow 转折判断法:
以迭代集群数去执行 k-means,用误差平方和(MSE)作为失真(Distortion)准度。
from sklearn.cluster import KMeans
distortions = []
# 用迭代将 1~10 都试一次
for i in range(1, 11):
km = KMeans(
n_clusters=i,
n_init=10,
max_iter=300,
random_state=0
)
km.fit(X)
# 样本到它们最近的聚类中心的距离平方和(越小越好)
distortions.append(km.inertia_)
# 作图
import matplotlib.pyplot as plt
plt.plot(range(1, 11), distortions, marker='o')
plt.xlabel('Number of clusters')
plt.ylabel('Distortion')
plt.tight_layout()
plt.show()
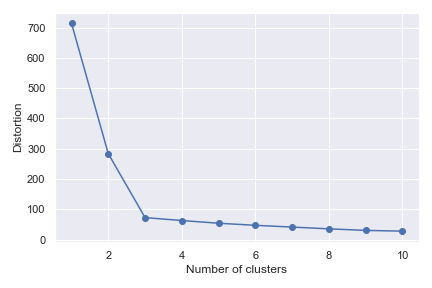
B2. Silhouette 轮廓系数法:
首先定义 Silhouette 轮廓如下:
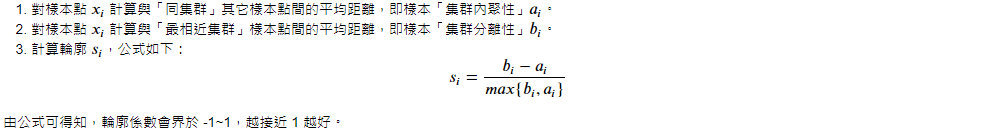
使用 sklearn 内建计算轮廓系数:
for i in range(2, 11):
km = KMeans(
n_clusters=i,
n_init=10,
max_iter=300,
random_state=0
)
km.fit(X)
y_pred = km.fit_predict(X)
print(f'{i:<2}, silhouette score: {silhouette_score(X, y_pred):.2f}')
>> 2 , silhouette score: 0.58
3 , silhouette score: 0.71
4 , silhouette score: 0.58
5 , silhouette score: 0.45
6 , silhouette score: 0.32
7 , silhouette score: 0.32
8 , silhouette score: 0.34
9 , silhouette score: 0.35
10, silhouette score: 0.35
可以发现当分群 3 时,轮廓系数最高。我们可以把它视觉化:
# k=3
from sklearn.cluster import KMeans
km = KMeans(n_clusters=3, n_init=10, max_iter=300, random_state=0)
y_pred = km.fit_predict(X)
import numpy as np
cluster_labels = np.unique(y_pred)
n_clusters = cluster_labels.shape[0]
from sklearn.metrics import silhouette_samples
sil = silhouette_samples(X, y_pred, metric='euclidean')
from matplotlib import cm
y_low, y_upp = 0, 0
yticks = []
# i: 1~3, c: 0~2
for i, c in enumerate(cluster_labels):
c_sil = sil[y_pred == c]
c_sil.sort()
y_upp += len(c_sil)
color = cm.jet(float(i) / n_clusters)
plt.barh(
range(y_low, y_upp),
c_sil,
height=1.0,
edgecolor='none',
color=color
)
yticks.append((y_low + y_upp) / 2.)
y_low += len(c_sil)
sil_avg = np.mean(sil)
plt.axvline(sil_avg, color="r", linestyle="--")
plt.yticks(yticks, cluster_labels + 1)
plt.ylabel('Cluster')
plt.xlabel('Silhouette coefficient')
plt.tight_layout()
plt.show()
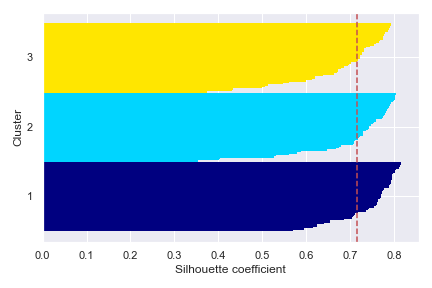
图片指标:
- 尽量让每个聚类样本数量相近
- 每个聚类基本上要大於轮廓系数(平均值)
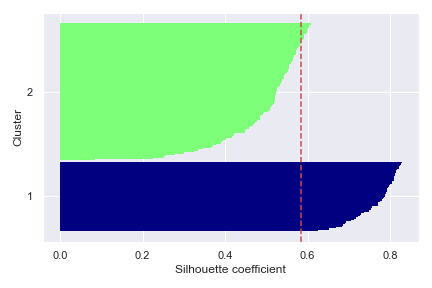
我们同时也可以比较 k=2,会发现其不符合上述指标。
6-2. Hierarchical Clustering 层次聚类
同为硬聚类的一种。将资料集逐次「合并或分裂」,直到要求的聚类数为止。
依照原理,层次聚类分成两种方法:
A. Divisive Hierarchical Clustering 分裂层次聚类
将所有样本视为一个聚类,将其逐步分割,直到达成指定聚类数为止。
B. Agglomerative Hierarchical Clustering 凝聚层次聚类
反之,以每个样本为聚类,不停将相近的聚类合并,直到达成指定聚类数为止。
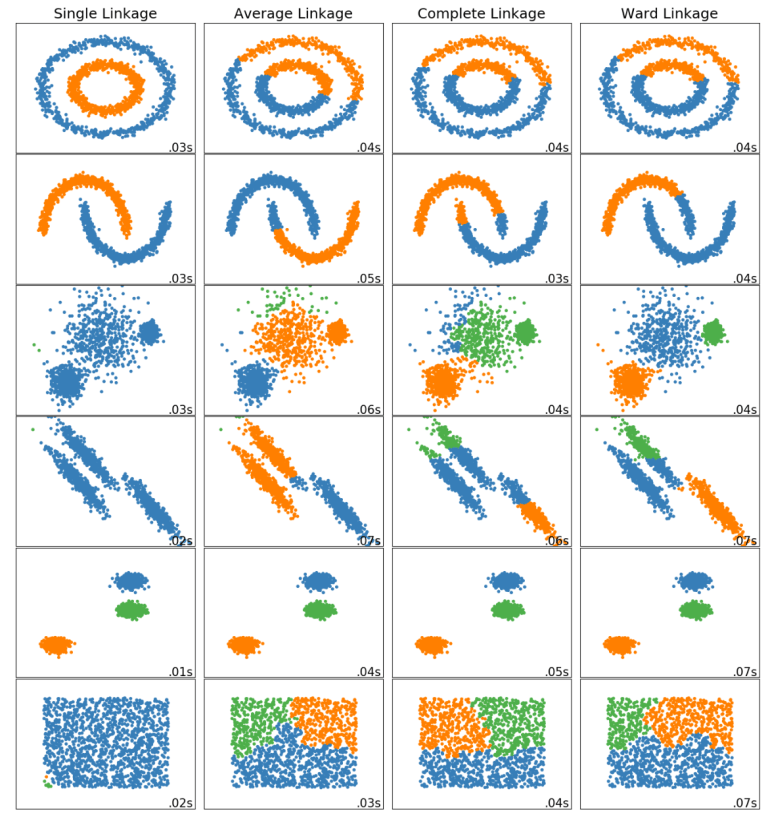
凝聚层次聚类又根据对「聚类间距离」定义不同,分以下四种算法:
Single Linkage 单一联动
聚类间距定义:不同群聚中最近两点间的距离。
特点:群聚的过程中产生「大者恒大」的效果,对离群很敏感。
Complete Linkage 完整联动
聚类间距定义:不同群聚中最远两点间的距离。
特点:比较容易产生「齐头并进」的效果,对离群很敏感。
Average Linkage 平均联动
聚类间距定义:不同聚类间点与点间的距离总和,取平均。
特点:比较容易产生「齐头并进」的效果,较不受离群影响。
Ward's method 沃德法
聚类间距定义:两群合并後,聚类中各点到聚类重心的距离平方和。
觉得抽象的化,用 sklearn 内建的图片来看就很易懂了:
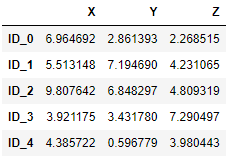
我们用固定乱数 np.random.seed 产生作范例:
import pandas as pd
import numpy as np
np.random.seed(123)
variables = ['X', 'Y', 'Z']
labels = ['ID_0', 'ID_1', 'ID_2', 'ID_3', 'ID_4']
X = np.random.random_sample([5, 3])*10
df = pd.DataFrame(X, columns=variables, index=labels)
df
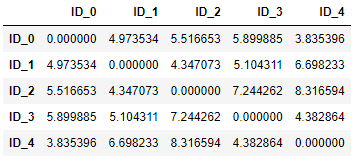
使用 pdist 计算点到点距离:
from scipy.spatial.distance import pdist, squareform
# pdist(X, metric=)
# X: nd-array,为 m*n 矩阵。
# metric: 距离求解法。默认 'euclidean'。
# 求解距离的函数。返回 Y 为 Cm取2 个观察值之间的成对距离。
dis_vec = pdist(df, metric='euclidean')
dis_vec
>> array([4.973534 , 5.51665266, 5.89988504, 3.83539555, 4.34707339,
5.10431109, 6.69823298, 7.24426159, 8.31659367, 4.382864 ])
把每个点到点的间距用矩阵表示:
# squareform: 将向量形式的距离表示转换成矩阵形式,或把矩阵形式转为向量形式。
matrix = squareform(dis_vec)
row_dis = pd.DataFrame(
matrix,
columns=labels,
index=labels
)
row_dis
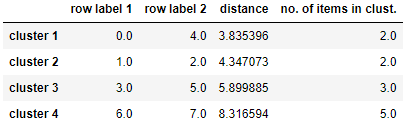
Likage(此处使用'complete'算法)
from scipy.cluster.hierarchy import linkage
row_clusters = linkage(df.values, method='complete', metric='euclidean')
pd.DataFrame(
row_clusters,
columns=['row label 1', 'row label 2', 'distance', 'no. of items in clust.'],
index=['cluster %d' % (i + 1) for i in range(row_clusters.shape[0])]
)
# 作图
import matplotlib.pyplot as plt
from scipy.cluster.hierarchy import dendrogram
row_dendr = dendrogram(
row_clusters,
labels=labels,
)
plt.tight_layout()
plt.ylabel('Euclidean distance')
plt.show()
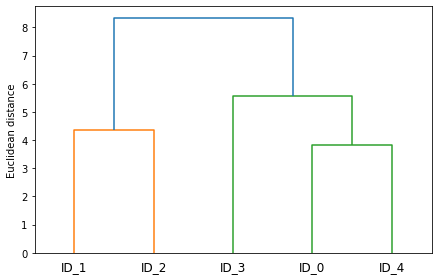
如果只想要得知分群结果,可用 sklearn 内建函数:
from sklearn.cluster import AgglomerativeClustering
ac = AgglomerativeClustering(
n_clusters=3,
affinity='euclidean',
linkage='complete'
)
labels = ac.fit_predict(X)
print(f'Cluster labels: {labels}')
>> Cluster labels: [1 0 0 2 1]
超参数:
1. n_clusters: 将要分成几群。默认 2。
2. affinity: 用於计算链接的度量。默认 'euclidean'。
3. linkage: 使用哪个链接标准,'ward'(默认), 'complete', 'average', 'single'。
6-3. DBSCAN 基於密度的杂讯应用空间聚类
全名 Density-Based Spatial Clustering of Applications w/ Noise。
不同於 K-means 与 Hierarchical 藉距离定义聚类,DBSCAN 是以「密度」定义聚类。
开始前,我们需要定义两个参数,分别是:
ε (eps):由这个参数值为半径划出的圆型区域称为 ε-邻域。
minPts:构成高密度区域需要最少有几个点。
详细步骤:
- 随机抽一资料点,探索其 ε-邻域,若有足够点则建立一新聚类,称它为 Core Point。
- 若无发现足够的点,则标签它为 Noise Point。
- 即使点被标签为杂讯,若往後标签过程中被视为聚类的一员,则标签为 Border Point。
如下图,即使单看点 C 会将其标签为杂讯,但它同时也是点 A 聚类的一员。
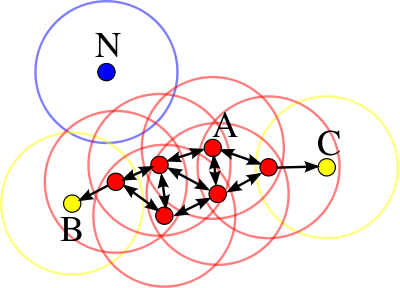
我们使用 make_moons 做范例:
from sklearn.datasets import make_moons
import matplotlib.pyplot as plt
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
plt.scatter(X[:, 0], X[:, 1], c='grey')
plt.tight_layout()
plt.show()
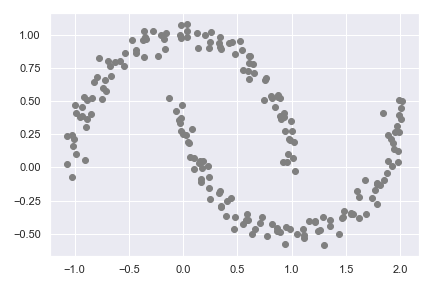
聚类方法:
# 用 K-means 做
from sklearn.cluster import KMeans, AgglomerativeClustering, DBSCAN
km = KMeans( n_clusters=2, random_state=0)
y_km = km.fit_predict(X)
# 用聚类分析做
ac = AgglomerativeClustering(n_clusters=2, affinity='euclidean', linkage='complete')
y_ac = ac.fit_predict(X)
# 用 DBSCAN 做
db = DBSCAN(eps=0.2, min_samples=5, metric='euclidean')
y_db = db.fit_predict(X)
超参数:
1. eps: 两个样本之间的最大距离,即 ε 。默认 0.5。
2. min_samples: 包括自身,将一个点视为核心点的邻近样本数(或总权重)。默认 5。
作图:
f, (ax1, ax2, ax3) = plt.subplots(1, 3, figsize=(12, 3))
axis = [ax1, ax2, ax3]
y_ = [y_km, y_ac, y_db]
titles = ['K-means clustering', 'Agglomerative clustering', 'DBSCAN']
for a, y, t in zip(axis, y_, titles):
a.scatter(
X[y == 0, 0], X[y == 0, 1],
c='lightblue', marker='o', s=40, label='cluster 1'
)
a.scatter(
X[y == 1, 0], X[y == 1, 1],
c='red', marker='o', s=40, label='cluster 2'
)
a.set_title(t)
plt.legend()
plt.tight_layout()
plt.show()
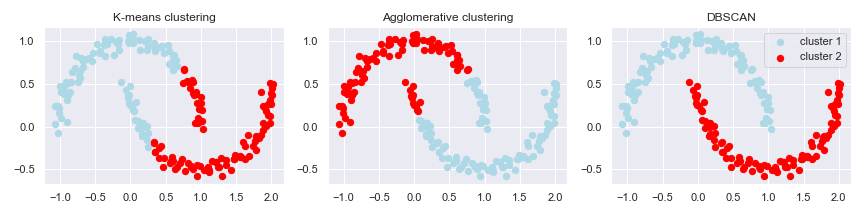
在聚类非线性资料集时,DBSCAN 表现通常比 K-means、Hierarchical 好。
最後,DBSCAN 也可以用来找寻资料集中的离群,请看 wine 范例:
from sklearn.datasets import load_wine
ds = load_wine()
X = pd.DataFrame(ds.data, columns=ds.feature_names)[['flavanoids', 'color_intensity']]
y = pd.DataFrame(ds.target, columns=['Wine'])
# 作图
plt.scatter(X.iloc[:, 0], X.iloc[:, 1], c='b', marker='o')
plt.xlabel('Concentration of flavanoids', fontsize=16)
plt.ylabel('Color intensity', fontsize=16)
plt.title('Concentration of flavanoids vs Color intensity', fontsize=20)
plt.show()
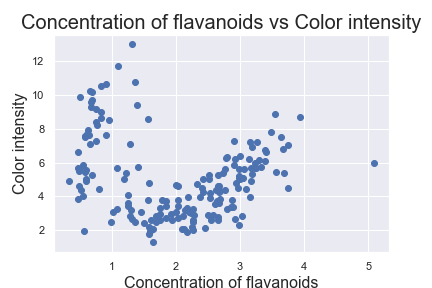
要注意的是这边只把影响最大的因子 'flavanoids' 丢进 DBSCAN
X_data = X.iloc[:, 0:1].values
from sklearn.cluster import DBSCAN
db = DBSCAN(eps=0.2, min_samples=19).fit(X_data)
import numpy as np
y_pred = db.labels_
# 作图
import matplotlib.pyplot as plt
plt.scatter(X.iloc[:, 0], X.iloc[:, 1], c=y_pred, marker='o')
plt.xlabel('Concentration of flavanoids', fontsize=16)
plt.ylabel('Color intensity', fontsize=16)
plt.show()
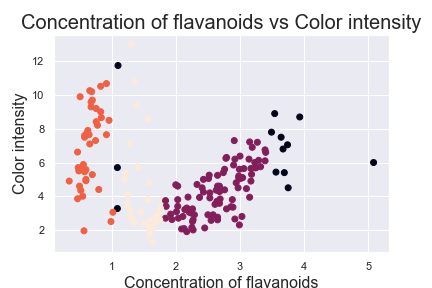
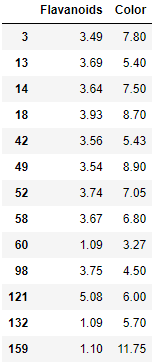
寻找 Outlier:
df[y_pred == -1]
至此,大致上说完 Cluster analysis 较常用的三种方法了。
结论:
- K-means 较有效率,但遇到大资料与非线性资料效果较差。
- Hierarchical 步骤为慢慢整并各类,在视觉化上较直观。
- DBSCAN 在寻找离群值 & 非线性资料时效果好,但在密度相近的异群间效果差。
.
.
.
.
.
补充资料
若有需求,可到以下网站习得更多:
<<: 不花一分钱加强WiFi安全:爲Router免费升级WPA3加密、用得更安心
如何从0开始当讲师-笔记
出处来自FB畅哥-如何从0开始当讲师 主讲者:孙治华 每个人能当讲师吗? 首先要看讲师的类型 EX:...
Day-4 Excel消失的0在哪里?
今天我们来更深入的探讨Excel中,首先先来介绍「日期」这个东西,并且我相信你也有遇过再Excel中...
DAY25-问答页面设计
前言: 几个基本的页面都设计得差不多啦~这次的挑战也快接近尾声了!今天就让阿森来介绍一个有点小特效...
Day29 参加职训(机器学习与资料分析工程师培训班),Tensorflow.keras & Pytorch
上午: Python机器学习套件与资料分析 使用tensorflow.keras 测试不同optim...
Day 22 : 模型优化 - 知识蒸馏 Knowledge Distillation
什麽是知识蒸馏 Knowledge Distillation 知识蒸馏 Knowledge Dist...