我们的基因体时代-AI, Data和生物资讯 Day20-注释基因资讯的BED档案格式和bedtools
上一篇我们的基因体时代-AI, Data和生物资讯 Day19-分析和处理基因变异的档案格式VCF的工具上一篇介绍在处理VCF档案时,可以用的工具,先从最简单的文字处理工具,如在命令行中的cut, grep, sed, awk,接者往下比较复杂的处理则可以使用bedtools, bcftools, vcftools来做运用。
再开始处理比较复杂的基因数据,就必须要先介绍相关跟基因数据相关的注释资料格式,可以参考圣塔克鲁兹加利福尼亚大学(University of California, Santa Cruz)的基因体浏览器网站(UCSC Genome Browser)之页面。
不过为何是圣塔克鲁兹加利福尼亚大学的基因体浏览器网站呢?
UCSC genome browser和基因注释相关的资料格式有什麽关系呢?这边就要提到一点点历史和国外生物资讯研究的风气,相对於国内生物医学的研究,国外往往讲求研究资料的共享,就像是美国国家生物技术资讯中心(National Center for Biotechnology Information)已经是目前世界上最重要的生物资料库,当人类基因体计画的开始,其实美国国家生物技术资讯中心就开始把相关资料置放在网站上供世界各地的科学家使用,当时人类基因组的资料库就是置放在NCBI资料库GeneBank之中,而除了美国的NCBI资料库外,欧洲也有另一个生物资讯中心叫做欧洲分子生物学实验室(European Molecular Biology Laboratory, EMBL),也建立了一个重要的公开资料库Ensembl,而当时圣塔克鲁兹加利福尼亚大学(University of California, Santa Cruz)的研究人员便决定也建立一套相关网站,来提供视觉化以及相关基因数据的浏览功能,当时在2000初的版本外观如下:
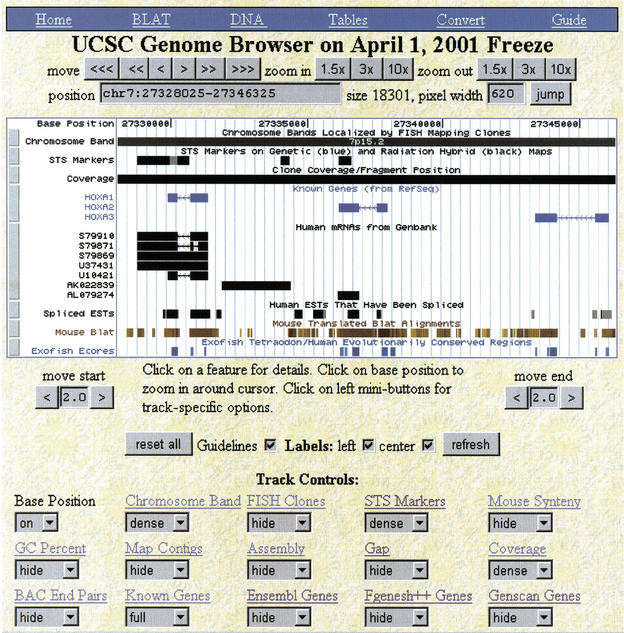
当时,这个网站就叫做UCSC Genome Browser,其初始的视觉化代码其实就是用来呈现线虫(C. elegans)定序资料中,基因模型的一个C语言的程序,其可以把资料呈现如下:
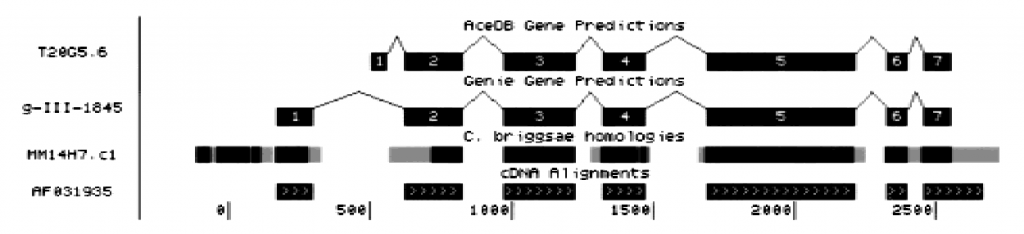
这样的目的是为了提供科学家一个更好理解基因序列资讯的方式,我想正常人类应该无法从单纯的一大串ATCGCGCTAGCTAGCTA中直接理解它的意义,所以基因组浏览器的目的就是提供一个有生物意义层次的架构:从染色体、外显子、基因、代谢路径等等层次去让生物学家来浏览这些基因序列资料。
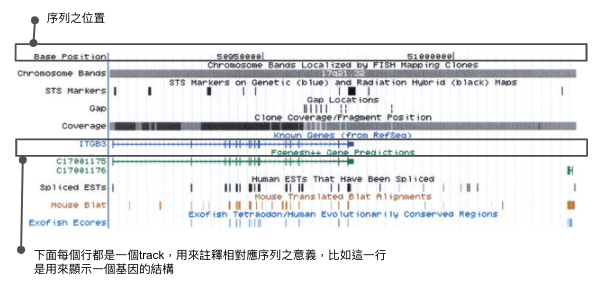
为了呈现这种注释效果,当时就使用所谓的track的模式,如同上面这张图,每一个track之间共通的关系就是在序列上的位置,然後其本身可以叠加不同意义,比如上图中圈起来的track,就是用来注释一个基因的结构,哪边是外显子,哪些是内显子,而如今这个方法已成为基因注释的主流方式,在UCSC Genome Browser在基因注解之track已经超过上千种了。
那呈现这样的视觉化资讯,要搭配相对应的资料格式,这时候就出现了所谓的BED档案格式(Browser Extensible Data),其便是为了对应这样视觉话需求的资料,当初最简单的版本就是3栏的数据,第一栏chrom:染色体名称,第二栏chromStart:起始位置(0-based index system),第三栏:chromEnd:结束位置(0-based index system),除了这三栏外,还有九个延伸的栏位:
- name:这笔资料的名称
- score:分数,可以是呈现时的色阶,介於0-100之间
- strand:所在为正股还是负股
- thickStart:呈现粗线条的起始
- thickEnd:呈现粗线条的结束
- itemRgb:颜色色阶
- blockCount:这区域的外显子数量
- blockSizes:外显子的大小
- blockStarts:每个外显子的起始位置
所以目前一份BED格式的资料会长的如下:
track name="ItemRGBDemo" description="Item RGB demonstration" visibility=2 itemRgb="On"
chr7 127471196 127472363 Pos1 0 + 127471196 127472363 255,0,0
chr7 127472363 127473530 Pos2 0 + 127472363 127473530 255,0,0
chr7 127473530 127474697 Pos3 0 + 127473530 127474697 255,0,0
chr7 127474697 127475864 Pos4 0 + 127474697 127475864 255,0,0
chr7 127475864 127477031 Neg1 0 - 127475864 127477031 0,0,255
chr7 127477031 127478198 Neg2 0 - 127477031 127478198 0,0,255
chr7 127478198 127479365 Neg3 0 - 127478198 127479365 0,0,255
chr7 127479365 127480532 Pos5 0 + 127479365 127480532 255,0,0
chr7 127480532 127481699 Neg4 0 - 127480532 127481699 0,0,255
使用bedtools来处理BED档案
BED档案,其实就是一群定义好的区域,这区域在基因体上有相对应的位置,那这时候就可以用很多工具来做处理,比如bedtools,这软件的解说页很清楚的表达了他怎麽处理不同之BED档案:
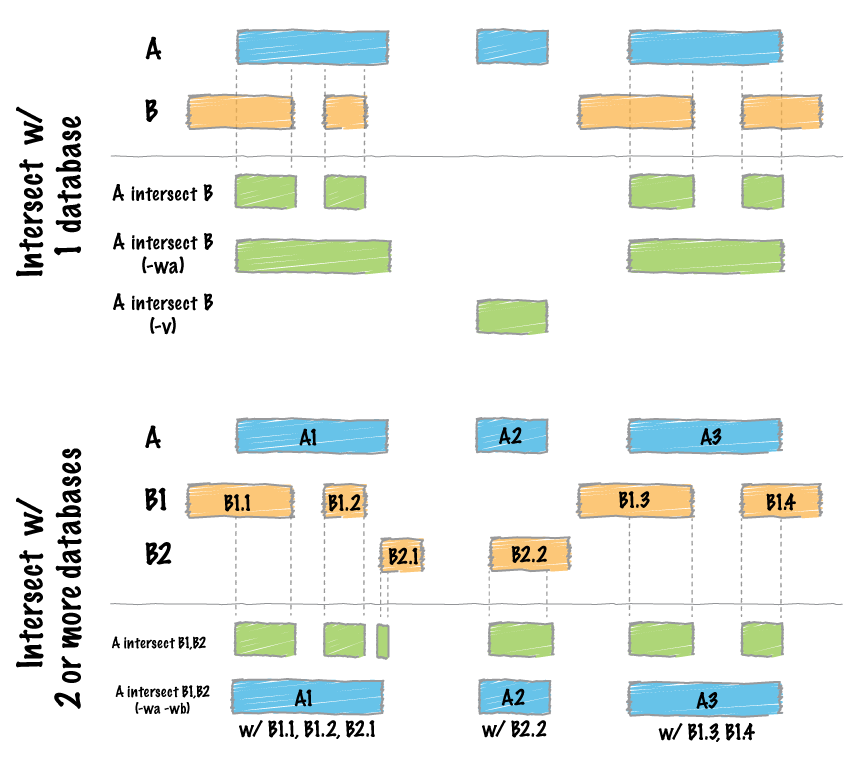
怎麽把这样的操作转换成生物意义来解释呢?
- 我们想要在一个变异点资料库中筛选出只在100个基因区域的变异点出来
- 我们想要筛选出在23对染色体上,只出现在外显子区域的变异点
- 我们想要找出位在调控BRCA相关的转录因子上面之变异点
- 我们想要看厂商A建库试剂的区域可以定序出哪些基因出来
- 我们手上有一个跟COVID感染相关的基因,想看在某一个病人身上的变异区域是否刚好位在这个区域中
当然,bedtools本身有很多功能,目前有41个函数,这些函数可以区分为几类功能,如基本区域筛选(intersect, window, closest, coverage, mapgenomecov, merge, cluster, complement, shift, subtract, slop, flank, sort, random, shuffle, sample, spacing, annotate)、多档案比对(multiinter, unionbedg)、格式转换(bamtobed, bedtobam, bamtofastq, bedpetobam, bed12tobed6)、BAM处理(multucov, tag, pairtobed, pairtopair)、统计分析(jaccard, reldist, fisher)、杂类(overlap, igv, linkns, makewindows, groupby, expand, split, summary)。
参考阅读:
Kent WJ, Sugnet CW, Furey TS, et al. The human genome browser at UCSC. Genome Res. 2002;12(6):996-1006. doi:10.1101/gr.229102
User-friendly, scalable tools and workflows for single-cell RNA-seq analysis. Nat Methods. 2021 Apr;18(4):327-328. doi: 10.1038/s41592-021-01102-w. PMID: 33782609; PMCID: PMC8299072.
Towards complete and error-free genome assemblies of all vertebrate species. Nature. 2021 Apr;592(7856):737-746. doi: 10.1038/s41586-021-03451-0. Epub 2021 Apr 28. PMID: 33911273; PMCID: PMC8081667.
Speir ML, Bhaduri A, Markov NS, Moreno P, Nowakowski TJ, Papatheodorou I, Pollen AA, Raney BJ, Seninge L, Kent WJ, Haeussler M. UCSC Cell Browser: Visualize Your Single-Cell Data. Bioinformatics. 2021 Jul 9:btab503. doi: 10.1093/bioinformatics/btab503. Epub ahead of print. PMID: 34244710.
Gonzalez JN, Zweig AS, Speir ML, Schmelter D, et al. The UCSC Genome Browser database: 2021 update Nucleic Acids Res. 2021 Jan 8;49(D1):D1046-D1057. PMID: 33221922 PMCID: PMC7779060
这个月的规划贴在这篇文章中我们的基因体时代-AI, Data和生物资讯 Overview,也会持续调整!我们的基因体时代是我经营的部落格,如有对於生物资讯、检验医学、资料视觉化、R语言有兴趣的话,可以来交流交流!
<<: 使用 Line Messaging Api 取得 User Profile
Day26 指派角色给使用者
昨天角色的 CRUD 功能都完成了,接着就是要把角色指派给使用者了,先建立一个 ViewModel ...
Golang-排序演算法
这篇文章算是做个纪录 把工作上遇到的问题,想到其他的解法记录下来 状况 资料因为从map取得,处理过...
予焦啦!基本的命令列
本节是以 Golang 上游 7ee4c1665477c6cf574cb9128deaf9d009...
开启Python IDLE
昨天我们下载完Python了,那我今天就来教大家怎麽使用吧! 首先我们可以在应用程序那边直接找P开头...
【Day30】最後一天的回顾笔记:我们没做到,但也做到了!
仔细回想一下,大概是在许多年前,就曾陆续看过 iT 邦帮忙相关广告或分享。但从没想过,这个品牌有一天...