我们的基因体时代-AI, Data和生物资讯 Day23- 基因注释资料在Bioconductor中视觉化之呈现:Gviz
上一篇我们的基因体时代-AI, Data和生物资讯 Day22- 基因注释资料在Bioconductor中的物件:AnnotationHub在示范间接GenomicRanges的使用前,先介绍一下怎麽在R环境下使用Bioconductor里面的资料包来取得跟人类基因体相关资讯的内容,以便进一步介绍GenomicRanges的进阶用法,那这边往下带到可以使用一些工具来视觉化这类的资讯,这边介绍Gviz。
我们延续上一篇的内容,先使用org.Hs.eg.db和hg19及hg38的资料来搜集跟血型基因RHD相关的资讯,想必大家可能多少都听过所谓的Rh血型,常常电视剧里面会提到Rh阴性血非常少见,尤其当Rh阴性孕妇怀了Rh阳性,比如最近火红的韩剧机智医师里面就刚好有这个案例,Rh血型阳性在台湾其实是指其具有RhD基因,其会产生所谓的RhD抗原,这个RhD基因就是我们这边想要来当作例子。
# load library
library(org.Hs.eg.db)
library(TxDb.Hsapiens.UCSC.hg38.knownGene)
library(TxDb.Hsapiens.UCSC.hg19.knownGene)
library(Gviz)
TxDb.hg38 <- TxDb.Hsapiens.UCSC.hg38.knownGene
TxDb.hg19 <- TxDb.Hsapiens.UCSC.hg19.knownGene
# use AnnotationHub to query gene ID
gene.inform.df <- AnnotationDbi::select(org.Hs.eg.db,
keys = c("RHD"),
keytype = c("SYMBOL"),
columns = c("ENTREZID"))
# use previous acquire gene ID inform to query gene annotation data
# Compare version hg19 and version hg38
hg19.cd.range.inform.df <- AnnotationDbi::select(TxDb.hg19,
keys = gene.inform.df$ENTREZID,
keytype = c("GENEID"),
columns = c("CDSCHROM", "CDSSTART","CDSID",
"CDSEND", "CDSID","CDSSTRAND")) %>%
mutate(WIDTH=CDSEND-CDSSTART)
hg38.cd.range.inform.df <- AnnotationDbi::select(TxDb.hg38,
keys = gene.inform.df$ENTREZID,
keytype = c("GENEID"),
columns = c("CDSCHROM", "CDSSTART","CDSID",
"CDSEND", "CDSID","CDSSTRAND")) %>%
mutate(WIDTH=CDSEND-CDSSTART)
利用基因注释资料建立IRange, GRanges物件
从上上篇的我们的基因体时代-AI, Data和生物资讯 Day21- 基因注释资料在Bioconductor中物件:IRanges和GenomicRanges内有介绍怎麽在R的环境下利用Bioconductor的两个专门用来建立基因资讯资料结构的包IRange和GenomicRanges,这边则是近一步直接用来自於人类基因组资料包取得的资讯来建立物件,以便进一步做分析和比较。
# construct IRange from annotationHub acquired result
hg19.Bloodtype_IRange <- IRanges::IRanges(start = hg19.cd.range.inform.df$CDSSTART,
width = hg19.cd.range.inform.df$WIDTH,
names = hg19.cd.range.inform.df$CDSID)
hg38.Bloodtype_IRange <- IRanges::IRanges(start = hg38.cd.range.inform.df$CDSSTART,
width = hg38.cd.range.inform.df$WIDTH,
names = hg38.cd.range.inform.df$CDSID)
# construct GRanges from IRanges
hg19.Bloodtype_GRanges <- GRanges(seqnames = Rle(hg19.cd.range.inform.df$CDSCHROM),
ranges = hg19.Bloodtype_IRange,
strand = Rle(hg19.cd.range.inform.df$CDSSTRAND),
geneid = hg19.cd.range.inform.df$GENEID,
length = hg19.cd.range.inform.df$WIDTH )
hg38.Bloodtype_GRanges <- GRanges(seqnames = Rle(hg38.cd.range.inform.df$CDSCHROM),
ranges = hg38.Bloodtype_IRange,
strand = Rle(hg38.cd.range.inform.df$CDSSTRAND),
geneid = hg38.cd.range.inform.df$GENEID,
length = hg38.cd.range.inform.df$WIDTH )
使用Gviz来做视觉化基因组资讯
在处理这类基因资讯,其实视觉化是很重要的工具,可以快速理解一些资料间的差异,比如这边的代码便是用来显示如何快速里用Gviz包来画基因资讯图,而下面就是用来建立最後要用plotTracks函数画图前,要建立的画轨。
# use Gviz to visualize the gene range data
axTrack <- GenomeAxisTrack()
hg19.bloodTypeTrack <- AnnotationTrack(hg19.Bloodtype_GRanges,
name='hg19 RHD gene',
genome='hg19')
hg38.bloodTypeTrack <- AnnotationTrack(hg38.Bloodtype_GRanges,
name='hg38 RHD gene',
genome='hg38')
hg38.idxTrack <- IdeogramTrack(genome="hg38", chromosome="chr1", name='chr1')
hg19.idxTrack <- IdeogramTrack(genome="hg19", chromosome="chr1", name='chr1')
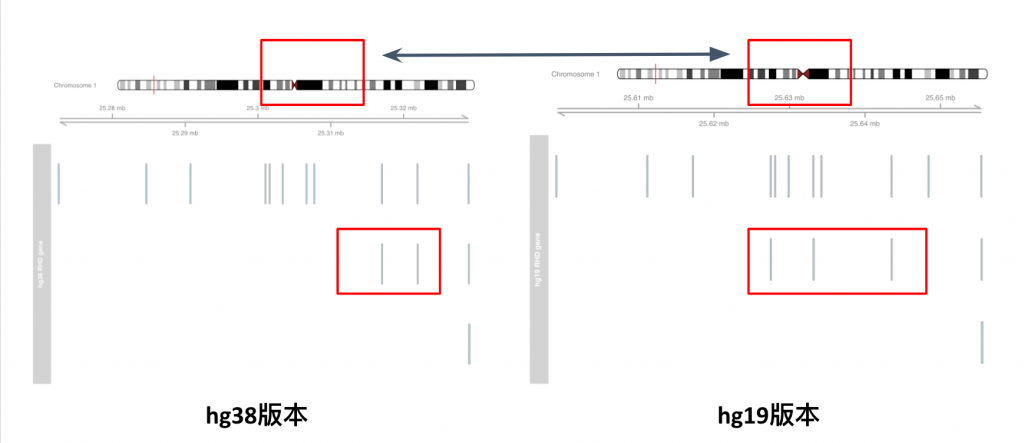
一览无遗的比较新旧版本基因组资料上RhD的差异
# plot hg38 version
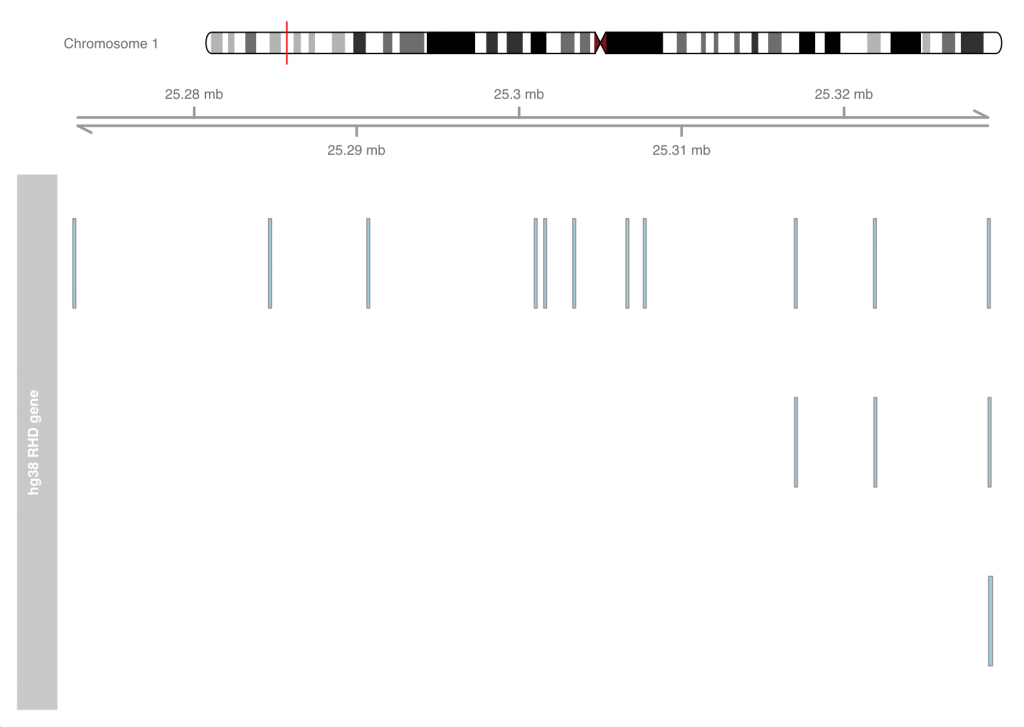
plotTracks(list(hg38.idxTrack, axTrack, hg38.bloodTypeTrack))
# plot hg19 version
plotTracks(list(hg19.idxTrack, axTrack, hg19.bloodTypeTrack))
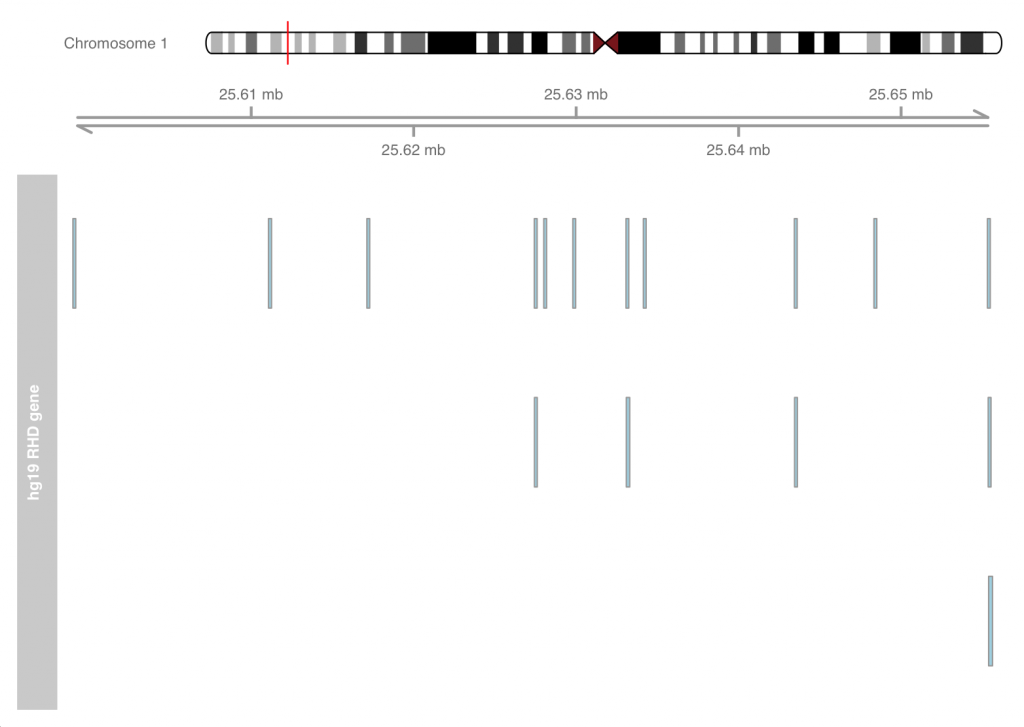
这样其实马上就可以看出在不同版本的基因资讯中,在RhD基因的基因转录区域资讯其实是有变化的,另外,本质上第一号染色体其实肉眼看也能发现不同,藉由这类视觉化其实能大幅帮助处理相关生物数据的能力!
接下来再继续深化怎麽使用Bioconductor内的工具来使用、分析和视觉化相关基因的资讯!
阅读参考
Chapter 16. Visualizing Genomic Data Using Gviz and Bioconductor. Statistical Genomics. 2016
Ou J, Zhu LJ (2019). “trackViewer: a Bioconductor package for interactive and integrative visualization of multi-omics data.” Nature Methods, 16, 453–454. doi: 10.1038/s41592-019-0430-y, https://doi.org/10.1038/s41592-019-0430-y.
这个月的规划贴在这篇文章中我们的基因体时代-AI, Data和生物资讯 Overview,也会持续调整!我们的基因体时代是我经营的部落格,如有对於生物资讯、检验医学、资料视觉化、R语言有兴趣的话,可以来交流交流!
<<: 那些被忽略但很好用的 Web API / DesignMode
EP16 - [TDD] 建立 Order 参数 (2/2)
Youtube 频道:https://www.youtube.com/c/kaochenlong ...
[Day 12] tinyML开发框架(一):TensorFlow Lite Micro初体验(上)
说到tinyML不得不说起「TinyML Machine Learning with TensorF...
Day27-好用的网页服务器-nginx(三)
前言 昨天的文章讲完前端 Nginx 的写法後,今天就要来进入後端的写法啦!在昨天的小结提到後端的写...
[PoEAA] Data Source Architectural Pattern - Data Mapper
本篇同步发布於个人Blog: [PoEAA] Data Source Architectural P...
{Day27}CameraX
CameraX CameraX提供了API,可在大多数Android设备上使用。 ProcessCa...